Technologieplattform Lichtmikroskopie
Die Technologieplattform Lichtmikroskopie (TPLM) betreibt seit 2022 sowohl high-end Lichtmikroskope als auch Ressourcen für Bildanalyse und entwickelt neue Technologien für die Lichtmikroskopie.
Die TPLM bietet Zugang zu Lichtmikroskopen am Centre for Structural Systems Biology (CSSB) in der Core Facility Advanced Light and Fluorescence Microscopy (ALFM) und am Fachbereich Biologie mit der Lichtmikroskopieplattform am Institut für Pflanzenwissenschaften und Mikrobiologie (IPM) sowie dem Institut für Zell- und Systembiologie der Tiere (IZS).
Mission
Die Mission der Technologieplattform Lichtmikroskopie (TPLM) ist es, Forschenden zu ermöglichen, für ihre Forschungsprojekte das volle Methodenspektrum der modernen Lichtmikroskopie zu nutzen. Um diese Mission zu erfüllen, hat die TPLM die folgenden Aufgaben:
- Infrastruktur: Die TPLM bietet Zugang zu moderner Lichmikroskopieinfrastruktur und Ressourcen zur Bildanalyse und erneuert und erweitert dafür regelmäßig ihre Geräteaustattung.
- Training und Unterstützung: Die TPLM unterstützt Forschende bei der Auswahl der besten Methoden zur Bildgebung und Bildanalyse für ihre Studien, trainiert Forschende im Umgang mit den Geräten der TPLM, unterstützt Forschende dabei Probleme zu lösen, die während laufenden Projekten auftreten, und führt gemeinschaftliche Projekte mit Forschungsgruppen durch.
- Methodenentwicklung: Die TPLM entwickelt neue Methoden und Technologien für die Bildgebung, welche von Nutzen für Forschende am Campus sind.
- Lehre: Die TPLM führt Lehrveranstaltungen zu den Grundlagen der Lichtmikroskopie und zu fortgeschrittenen Lichtmikroskopietechniken durch.
Welche Unterstützung bietet die TPLM?
Die TPLM bietet verschiedene Arten der bedarfsorientierten Unterstützung an:
- Unabhängige Nutzung von Geräten nach entsprechendem Training
- Begleitende Unterstützung von Projekten
- Gemeinsame Entwicklung und Durchführung von Projekten im Bereich Lichtmikroskopie
Wir unterstützen Forschende auch bei der Verarbeitung und der quantitativen Analyse von Lichtmikroskopiedaten.
Kontakt und Zugang zu den Geräten
Die TPLM bietet derzeit Zugang zu Lichtmikroskopen in der ALFM Facility am CSSB. Die ALFM Facility ist auch zugänglich für externe Nutzer:innen.
Das Team der Technologieplattform Lichtmikroskopie unterstützt Sie gerne bei der Planung und Durchführung lichtmikroskopischer Experimente und bei der Analyse lichtmikroskopischer Bilddaten. Bitte kontaktieren Sie den Plattformkoordinator Roland Thünauer für weitere Informationen.
Standorte
Centre for Structural Systems Biology (CSSB)
c/o Deutsches Elektronen-Synchrotron DESY
Notkestraße 85, Gebäude 15
22607 Hamburg
Lichtmikroskopieplattform am Fachbereich Biologie
Institut für Pflanzenwissenschaften und Mikrobiologie (IPM)
Ohnhorststr. 18
22609 Hamburg
Institut für Zell- und Systembiologie der Tiere (IZS)
Martin-Luther-King-Platz 3
20146 Hamburg
Wie kann ich die Geräte nutzen?
Für neue Nutzer:innen wird zuerst ein Treffen vereinbart, bei dem besprochen wird, wie die TPLM das Projekt am besten unterstützen kann und wie Geräte der TPLM genutzt werden können.
Bitte wenden Sie sich per E-Mail an Roland Thünauer, um ein Treffen zu vereinbaren oder um mehr Informationen zu erhalten.
Wie kann ich Geräte buchen?
An der ALFM Core Facility am CSSB Hamburg
Geräte an der ALFM Facility sind buchbar über den PPMS Buchungskalender. Der Zugang zum Buchungssystem wird nach einem Training und dem Unterzeichnen der Nutzungsbedingungen (PDF) eingerichtet. Weitere Informationen befinden sich auf der Website der ALFM Facility.
Am Fachbereich Biologie
Für Informationen und Fragen zum Fachbereich Biologie wenden Sie sich bitte an Roland Thünauer.
Projekte
Die TPLM ist offen für Kooperationsprojekte mit Forschenden mit besonderem Fokus auf Methodenentwicklung für die Lichtmikroskopie.
Aktuelle Projekte
CUI: AIM Projekt cryoLM
In diesem Projekt werden neue Beleuchtungsschemata für Licht-Kryo-Mikroskopie (cryoLM) entwickelt.
Kooperationspartner:
Kay Grünewald, Franz Kärtner, Kartik Ayyer, Rainer Kaufmann und Felix Lehmkühler
Publikationen
Wie kann in Publikationen angegeben werden, dass die TPLM genutzt wurde?
Wenn Geräte oder die Unterstützung der Technologieplattform Lichtmikroskopie in Anspruch genommen wurden, bitten wir alle Nutzer:innen der TPLM darum, dies in ihren Publikationen anzugeben.
Der folgende Text kann als Vorlage für den Acknowledgement-Abschnitt eines Manuskripts dienen:
We thank the Technology Platform Light Microscopy (TPLM) at Universität Hamburg (UHH), in particular XXX, for support with light microscopy image acquisition and analysis.
Publikationen mit Beteiligung der TPLM
2023
A TriPPPro-Nucleotide Reporter with Optimized Cell-Permeable Dyes for Metabolic Labeling of Cellular and Viral DNA in Living Cells.
Sterrenberg VT, Stalling D, Knaack JIH, Soh TK, Bosse JB, Meier C.
Angew Chem Int Ed Engl. 2023; 62(38):e202308271. doi: 10.1002/anie.202308271.
PMID: 37435767
Kupffer cell-like syncytia replenish resident macrophage function in the fibrotic liver.
Peiseler M, Araujo David B, Zindel J, Surewaard BGJ, Lee WY, Heymann F, Nusse Y, Castanheira FVS, Shim R, Guillot A, Bruneau A, Atif J, Perciani C, Ohland C, Ganguli Mukherjee P, Niehrs A, Thuenauer R, Altfeld M, Amrein M, Liu Z, Gordon PMK, McCoy K, Deniset J, MacParland S, Ginhoux F, Tacke F, Kubes P.
Science. 2023; 381(6662):eabq5202. doi: 10.1126/science.abq5202.
PMID: 37676943
Real-time monitoring of cell surface protein arrival with split luciferases.
Fischer AAM, Schatz L, Baaske J, Römer W, Weber W, Thuenauer R.
Traffic. 2023; 24(10):453-462. doi: 10.1111/tra.12908.
PMID: 37403269
Global analysis of putative phospholipases in Plasmodium falciparum reveals an essential role of the phosphoinositide-specific phospholipase C in parasite maturation.
Burda PC, Ramaprasad A, Bielfeld S, Pietsch E, Woitalla A, Söhnchen C, Singh MN, Strauss J, Sait A, Collinson LM, Schwudke D, Blackman MJ, Gilberger TW.
mBio. 2023; 14(4):e0141323. doi: 10.1128/mbio.01413-23.
PMID: 37489900
Comparative proteomics of vesicles essential for the egress of Plasmodium falciparum gametocytes from red blood cells.
Sassmannshausen J, Bennink S, Distler U, Küchenhoff J, Minns AM, Lindner SE, Burda PC, Tenzer S, Gilberger TW, Pradel G.
Mol Microbiol. 2023. doi: 10.1111/mmi.15125. Online ahead of print.
PMID: 37492994
A validated protocol to UV-inactivate SARS-CoV-2 and herpesvirus-infected cells.
Soh TK, Pfefferle S, Wurr S, von Possel R, Oestereich L, Rieger T, Uetrecht C, Rosenthal M, Bosse JB.
PLoS One. 2023; 18(5):e0274065. doi: 10.1371/journal.pone.0274065.
PMID: 37163509
Non-functional ubiquitin C-terminal hydrolase L1 drives podocyte injury through impairing proteasomes in autoimmune glomerulonephritis.
Reichelt J, Sachs W, Frömbling S, Fehlert J, Studencka-Turski M, Betz A, Loreth D, Blume L, Witt S, Pohl S, Brand J, Czesla M, Knop J, Florea BI, Zielinski S, Sachs M, Hoxha E, Hermans-Borgmeyer I, Zahner G, Wiech T, Krüger E, Meyer-Schwesinger C.
Nat Commun. 2023; 14(1):2114. doi: 10.1038/s41467-023-37836-8.
PMID: 37055432
CXCR5+PD-1++ CD4+ T cells colonize infant intestines early in life and promote B cell maturation.
Jordan-Paiz A, Martrus G, Steinert FL, Kaufmann M, Sagebiel AF, Schreurs RRCE, Rechtien A, Baumdick ME, Jung JM, Möller KJ, Wegner L, Grüttner C, Richert L, Thünauer R, Schroeder-Schwarz J, van Goudoever JB, Geijtenbeek TBH, Altfeld M, Pals ST, Perez D, Klarenbeek PL, Tomuschat C, Sauter G, Königs I, Schumacher U, Friese MA, Melling N, Reinshagen K, Bunders MJ.
Cell Mol Immunol. 2023; 20(2):201-213. doi: 10.1038/s41423-022-00944-4.
PMID: 36600048
Slow integrin-dependent migration organizes networks of tissue-resident mast cells.
Kaltenbach L, Martzloff P, Bambach SK, Aizarani N, Mihlan M, Gavrilov A, Glaser KM, Stecher M, Thünauer R, Thiriot A, Heger K, Kierdorf K, Wienert S, von Andrian UH, Schmidt-Supprian M, Nerlov C, Klauschen F, Roers A, Bajénoff M, Grün D, Lämmermann T.
Nat Immunol. 2023; 24(6):915-924. doi: 10.1038/s41590-023-01493-2.
PMID: 37081147
A Microtubule-Associated Protein Is Essential for Malaria Parasite Transmission.
Wichers-Misterek JS, Binder AM, Mesén-Ramírez P, Dorner LP, Safavi S, Fuchs G, Lenz TL, Bachmann A, Wilson D, Frischknecht F, Gilberger TW.
mBio. 2023; 14(1):e0331822. doi: 10.1128/mbio.03318-22.
PMID: 36625655
Adsorption and Inactivation of SARS-CoV-2 on the Surface of Anatase TiO2(101).
Kohantorabi M, Wagstaffe M, Creutzburg M, Ugolotti A, Kulkarni S, Jeromin A, Krekeler T, Feuerherd M, Herrmann A, Ebert G, Protzer U, Guédez G, Löw C, Thuenauer R, Schlueter C, Gloskovskii A, Keller TF, Di Valentin C, Stierle A, Noei H.
ACS Appl Mater Interfaces. 2023 doi: 10.1021/acsami.2c22078.
PMID: 36723177
Formation kinetics and physicochemical properties of mesoscopic Alpha-Synuclein assemblies modulated by sodium chloride and a distinct pulsed electric field.
Wang M, Thuenauer R, Schubert R, Gevorgyan S, Lorenzen K, Brognaro H, Betzel C.
Soft Matter. 2023 doi: 10.1039/d2sm01615j.
PMID: 36723049
Dimeric Lectin Chimeras as Novel Candidates for Gb3-Mediated Transcytotic Drug Delivery through Cellular Barriers.
Xu M, Antonova M, Salavei P, Illek K, Meléndez AV, Omidvar R, Thuenauer R, Makshakova O, Römer W.
Pharmaceutics. 2023 15(1):225. doi: 10.3390/pharmaceutics15010225.
PMID: 36678854
2022
Protocol for live-cell fluorescence-guided cryoFIB-milling and electron cryo-tomography of virus-infected cells.
Franken LE, Rosch R, Laugks U, Grünewald K.
STAR Protoc. 2022 3(4):101696. doi: 10.1016/j.xpro.2022.101696.
PMID: 36149798
MARVEL domain containing CMTM4 affects CXCR4 trafficking.
Bona A, Seifert M, Thünauer R, Zodel K, Frew IJ, Römer W, Walz G, Yakulov TA.
Mol Biol Cell. 2022 33(13):ar116. doi: 10.1091/mbc.E22-05-0152.
PMID: 36044337
Functional inactivation of Plasmodium falciparum glycogen synthase kinase GSK3 modulates erythrocyte invasion and blocks gametocyte maturation.
Alder A, Wilcke L, Pietsch E, von Thien H, Pazicky S, Löw C, Mesen-Ramirez P, Bachmann A, Burda PC, Kunick C, Sondermann H, Wilson D, Gilberger TW.
J Biol Chem. 2022 298(9):102360. doi: 10.1016/j.jbc.2022.102360.
PMID: 35961464
Nipah Virus Infection Generates Ordered Structures in Cellulo.
Vázquez CA, Widerspick L, Thuenauer R, Schneider C, Reimer R, Neira P, Olal C, Heung M, Niemetz L, Lawrence P, Kucinskaite-Kodze I, Redecke L, Escudero-Pérez B.
Viruses. 2022 14(7):1523. doi: 10.3390/v14071523.
PMID: 35891503
The Lectin LecB Induces Patches with Basolateral Characteristics at the Apical Membrane to Promote Pseudomonas aeruginosa Host Cell Invasion.
Thuenauer R, Kühn K, Guo Y, Kotsis F, Xu M, Trefzer A, Altmann S, Wehrum S, Heshmatpour N, Faust B, Landi A, Diedrich B, Dengjel J, Kuehn EW, Imberty A, Römer W.
mBio. 2022 13(3):e0081922. doi: 10.1128/mbio.00819-22.
PMID: 35491830
Human cytomegalovirus forms phase-separated compartments at viral genomes to facilitate viral replication.
Caragliano E, Bonazza S, Frascaroli G, Tang J, Soh TK, Grünewald K, Bosse JB, Brune W.
Cell Rep. 2022 38(10):110469. doi: 10.1016/j.celrep.2022.110469.
PMID: 35263605
Intermittent bulk release of human cytomegalovirus.
Flomm FJ, Soh TK, Schneider C, Wedemann L, Britt HM, Thalassinos K, Pfitzner S, Reimer R, Grünewald K, Bosse JB.
PLoS Pathog. 2022 18(8):e1010575. doi: 10.1371/journal.ppat.1010575.
PMID: 35925870
A non-reactive natural product precursor of the duocarmycin family has potent and selective antimalarial activity.
Alder A, Struck NS, Xu M, Johnson JW, Wang W, Pallant D, Cook MA, Rambow J, Lemcke S, Gilberger TW, Wright GD.
Cell Chem Biol. 2022 29(5):840-853.e6. doi: 10.1016/j.chembiol.2021.10.005.
PMID: 34710358
Geräteübersicht

Foto: UHH/Feuerböther
Analysecomputer (Platzhalter-Bild)
Beschreibung: Der Analysecomputer verfügt über 768 GB Arbeitsspeicher und zwei NVIDIA RTX A6000 Graphikkarten für die Bearbeitung und Analyse großer Bilddatenmengen. Auf dem Computer sind FIJI, Imaris, Arivis und Matlab als Bildanalysesoftware vorhanden.
Hauptanwendungen: Bildbearbeitung und quantitative Bildanalyse von Mikroskopiedaten

Foto: UHH/Feuerböther
Leica SP8 Konfokalmikroskop mit FALCON FLIM Einheit
Beschreibung: Dieses inverse Point-Scanning-Konfokalmikroskop von Leica ist ausgestattet mit 405 nm Laser, Weißlichtlaser und 4 Hybriddetektoren. Das Mikroskop verfügt über eine Inkubationseinheit für Lebendzellmessungen und eine FALCON FLIM Einheit. Das Mikroskop ist programmierbar über das Navigator-Modul der Leica LASX Software. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Konfokalmikroskopie, FLIM, FRET, Lebendzellmikroskopie

Foto: UHH/Feuerböther
Leica cryoCLEM Weitfeldmikroskop
Beschreibung: Dieses aufrechte Weitfeldmikroskop von Leica ist ausgestattet mit einer Kryo-Stage, um vitrifizierte Proben auf Elektronenmikroskopiegrids unter ständiger Kühlung mit flüssigem Stickstoff untersuchen zu können. Als Fluoreszenzanregungslichtquelle ist ein Quecksilber-Metall-Halid-Brenner (EL6000) vorhanden. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Licht-Kryo-Mikroskopie, Weitfeldmikroskopie, korrelative Licht- und Elektronen-Kryo-Mikroskopie

Foto: UHH/Feuerböther
Leica cryoCLEM Stellaris 8 Konfokalmikroskop
Beschreibung: Dieses aufrechte Konfokalmikroskop von Leica ist ausgestattet mit einer Kryo-Stage, um vitrifizierte Proben auf Elektronenmikroskopiegrids unter ständiger Kühlung mit flüssigem Stickstoff untersuchen zu können. Als Fluoreszenzanregungslichtquellen sind ein gepulster 405 nm Laser, ein gepulster Weißlichtlaser und 488 nm, 561 nm und 633 nm cw Laser vorhanden. Das Mikroskop ist mit vier Hybriddetektoren ausgestattet und hat eine FLIM-Einheit. Zusätzlich kann das Mikroskop im Weitfeldmodus betrieben werden und verfügt über eine LED-Fluoreszenzweitfeldanregung. Das Mikroskop ist programmierbar über das Navigator-Modul der Leica LASX Software. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Licht-Kryo-Mikroskopie, Konfokalmikroskopie, FLIM, FRET, Weitfeldmikroskopie, korrelative Licht- und Elektronen-Kryo-Mikroskopie
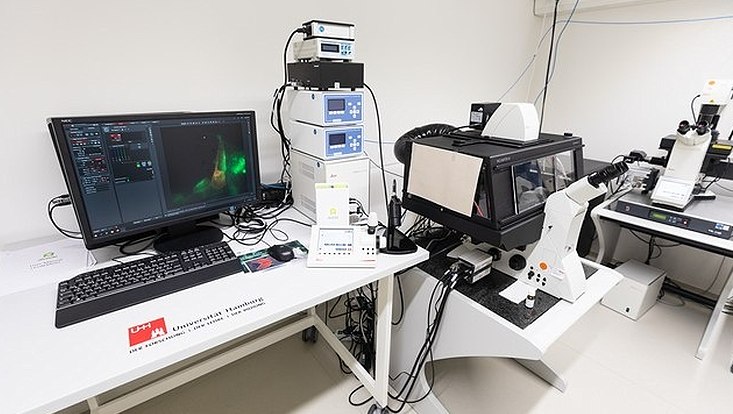
Foto: UHH/Feuerböther
Leica DMi8 inverses Weitfeldmikroskop für Lebendzellmessungen und mit Primo Alveole Micropatterning Einheit
Beschreibung: Dieses inverse Weitfeldmikroskop von Leica ist ausgestattet mit einer Inkubationseinheit für Lebendzellmessungen und einer LED-Fluoreszenzanregungslichtquelle. Zusätzlich ist eine Primo Alveole Micropatterning Einheit angebracht. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Lebendzellmikroskopie, Weitfeldmikroskopie, Phasenkontrastmikroskopie, DIC, Micropatterning

Foto: UHH/Feuerböther
Leica DMi8 inverses Weitfeldmikroskop für Lebendzellmessungen mit THUNDER Einheit
Beschreibung: Dieses inverse Weitfeldmikroskop von Leica ist ausgestattet mit einer Inkubationseinheit für Lebendzellmessungen, einer LED-Fluoreszenzanregungslichtquelle und der THUNDER Software für Computational Clearing. Das Mikroskop ist programmierbar für komplexe Aufgaben, wie beispielsweise das Abscannen von Multiwellplatten. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Lebendzellmikroskopie, Weitfeldmikroskopie, Phasenkontrastmikroskopie, DIC, Scannen von Multiwellplatten

Foto: UHH/Feuerböther
Leica DM6 B aufrechtes Weitfeldmikroskop #1
Beschreibung: Dieses aufrechte Weitfeldmikroskop von Leica ist ausgestattet mit einer Fluoreszenzanregungslichtquelle. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Weitfeldmikroskopie, Phasenkontrastmikroskopie, DIC, Histologie

Foto: UHH/Feuerböther
Leica DM6 B aufrechtes Weitfeldmikroskop #2
Beschreibung: Dieses aufrechte Weitfeldmikroskop von Leica ist ausgestattet mit einer Fluoreszenzanregungslichtquelle und ist voll motorisiert. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Weitfeldmikroskopie, Phasenkontrastmikroskopie, DIC, Histologie

Foto: UHH/Feuerböther
Leica DM6 B aufrechtes Weitfeldmikroskop #3
Beschreibung: Dieses aufrechte Weitfeldmikroskop von Leica ist ausgestattet mit einer Fluoreszenzanregungslichtquelle. Das Mikroskop im BSL2 Labor von Tim Gilberger am CSSB Hamburg untergebracht.
Hauptanwendungen: Weitfeldmikroskopie, Phasenkontrastmikroskopie, DIC, Histologie

Foto: UHH/Feuerböther
Leica SR GSD 3D Super-Resolution-Mikroskop
Beschreibung: Dieses inverse Mikroskop von Leica ist ausgestattet für Single Molecule Localization Microscopy (SMLM) Super-Resolution Mikroskopie und einer speziellen Stage, die direkt an das Objektiv gekoppelt ist. Die Fluoreszenzanregung kann im Weitfeldmodus und im Total Internal Reflection Fluorescence (TIRF)-Modus erfolgen. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Super-Resolution Mikroskopie, TIRF, STORM, DNA-PAINT

Foto: UHH/Feuerböther
Spinning-Disc- und SIM-Mikroskop mit FRAP Einheit
Beschreibung: Dieses inverse Mikroskop von Nikon hat mehrere Modi in denen es betrieben werden kann. Es ist eine Yokogawa CSU W1 Spinning Disc Einheit mit einer Andor iXon EMCCD Kamera vorhanden und eine Nikon E-SIM Einheit für Structured Illumination Microscopy (SIM). Darüber hinaus ist das Mikroskop mit einer Einheit für Fluorescence Recovery after Photobleaching (FRAP) von Rapp Optoelectronics ausgestattet. Es ist eine Inkubationseinheit für Lebendzellmessungen vorhanden. Das Mikroskop ist voll programmierbar über das JOBS-Modul in der Nikon NIS Elements Software. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Spinning-Disc-Konfokalmikroskopie, SIM, FRAP, Photoaktivierung, Lebendzellmikroskopie, Scannen von Multiwellplatten

Foto: UHH/Feuerböther
LSM880 Airyscan Konfokalmikroskop
Beschreibung: Dieses inverse Point-Scanning-Konfokalmikroskop von Zeiss hat einen Airyscandetektor, welcher genutzt werden kann, um eine ungefähr 1,8-fache Erhöhung der Auflösung gegenüber normalen Konfokalmikroskopen zu erreichen, und einen Spectral Imaging Detektor. Es ist mit einer Inkubationskammer für Lebendzellmessungen ausgestattet. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Konfokalmikroskopie, Lebendzellmikroskopie, Airyscan-Mikroskopie

Foto: UHH/Feuerböther
Elyra PS.1 Super-Resolution-Mikroskop
Beschreibung: Dieses inverse Super-Resolution-Mikroskop von Zeiss kann für Single Molecule Localization Microscopy (SMLM) Mikroskopie und für Structured Illumination Microscopy (SIM) verwendet werden. Die Fluoreszenzanregung kann im Weitfeldmodus oder im Total Internal Reflection Fluorescence (TIRF)-Modus erfolgen. Es ist mit einer Inkubationskammer für Lebendzellmessungen ausgestattet. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Super-Resolution Mikroskopie, TIRF, STORM, PALM, DNA-PAINT, SIM, Weitfeldmikroskopie

Foto: UHH/Feuerböther
Lattice-Light-Sheet-Mikroskop
Beschreibung: Das Lattice-Light-Sheet-Mikroskop von Zeiss ist schnelle volumetrische Aufnahmen von lebenden Zellen mit besonders geringer Phototoxizität und gleichzeitig hoher örtlicher Auflösung geeignet. Es verfügt über zwei Hamamatsu sCMOS Kameras. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Lattice-Light-Sheet Mikroskopie, Lebendzellmikroskopie

Foto: UHH/Feuerböther
BSL2 Zellkulturwerkbank und Inkubatoren
Beschreibung: Die Zellkulturwerkbank und zwei Inkubatoren sind voll ausgestattet damit Nutzende der ALFM-Facility ihre Lebendzellproben für die Mikroskopie vorbereiten und vorinkubieren können. Die Zellkulturwerkbank und die Inkubatoren sind in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Vorbereitung von Lebendzellproben für die Mikroskopie

Foto: UHH/Feuerböther
Leica M125C Stereomikroskop
Beschreibung: Das M125C ist ein kleines Stereomikroskop von Leica mit LED-Weißlichtquelle. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Inspektion und Präparation von Mikroskopieproben.

Foto: UHH/Feuerböther
Analysecomputer (Platzhalter-Bild)
Beschreibung: Der Analysecomputer verfügt über 768 GB Arbeitsspeicher und zwei NVIDIA RTX A6000 Graphikkarten für die Bearbeitung und Analyse großer Bilddatenmengen. Auf dem Computer sind FIJI, Imaris, Arivis und Matlab als Bildanalysesoftware vorhanden.
Hauptanwendungen: Bildbearbeitung und quantitative Bildanalyse von Mikroskopiedaten

Foto: UHH/Feuerböther
Leica SP8 Konfokalmikroskop mit FALCON FLIM Einheit
Beschreibung: Dieses inverse Point-Scanning-Konfokalmikroskop von Leica ist ausgestattet mit 405 nm Laser, Weißlichtlaser und 4 Hybriddetektoren. Das Mikroskop verfügt über eine Inkubationseinheit für Lebendzellmessungen und eine FALCON FLIM Einheit. Das Mikroskop ist programmierbar über das Navigator-Modul der Leica LASX Software. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Konfokalmikroskopie, FLIM, FRET, Lebendzellmikroskopie

Foto: UHH/Feuerböther
Leica cryoCLEM Weitfeldmikroskop
Beschreibung: Dieses aufrechte Weitfeldmikroskop von Leica ist ausgestattet mit einer Kryo-Stage, um vitrifizierte Proben auf Elektronenmikroskopiegrids unter ständiger Kühlung mit flüssigem Stickstoff untersuchen zu können. Als Fluoreszenzanregungslichtquelle ist ein Quecksilber-Metall-Halid-Brenner (EL6000) vorhanden. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Licht-Kryo-Mikroskopie, Weitfeldmikroskopie, korrelative Licht- und Elektronen-Kryo-Mikroskopie

Foto: UHH/Feuerböther
Leica cryoCLEM Stellaris 8 Konfokalmikroskop
Beschreibung: Dieses aufrechte Konfokalmikroskop von Leica ist ausgestattet mit einer Kryo-Stage, um vitrifizierte Proben auf Elektronenmikroskopiegrids unter ständiger Kühlung mit flüssigem Stickstoff untersuchen zu können. Als Fluoreszenzanregungslichtquellen sind ein gepulster 405 nm Laser, ein gepulster Weißlichtlaser und 488 nm, 561 nm und 633 nm cw Laser vorhanden. Das Mikroskop ist mit vier Hybriddetektoren ausgestattet und hat eine FLIM-Einheit. Zusätzlich kann das Mikroskop im Weitfeldmodus betrieben werden und verfügt über eine LED-Fluoreszenzweitfeldanregung. Das Mikroskop ist programmierbar über das Navigator-Modul der Leica LASX Software. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Licht-Kryo-Mikroskopie, Konfokalmikroskopie, FLIM, FRET, Weitfeldmikroskopie, korrelative Licht- und Elektronen-Kryo-Mikroskopie

Foto: UHH/Feuerböther
Leica DMi8 inverses Weitfeldmikroskop für Lebendzellmessungen und mit Primo Alveole Micropatterning Einheit
Beschreibung: Dieses inverse Weitfeldmikroskop von Leica ist ausgestattet mit einer Inkubationseinheit für Lebendzellmessungen und einer LED-Fluoreszenzanregungslichtquelle. Zusätzlich ist eine Primo Alveole Micropatterning Einheit angebracht. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Lebendzellmikroskopie, Weitfeldmikroskopie, Phasenkontrastmikroskopie, DIC, Micropatterning

Foto: UHH/Feuerböther
Leica DMi8 inverses Weitfeldmikroskop für Lebendzellmessungen mit THUNDER Einheit
Beschreibung: Dieses inverse Weitfeldmikroskop von Leica ist ausgestattet mit einer Inkubationseinheit für Lebendzellmessungen, einer LED-Fluoreszenzanregungslichtquelle und der THUNDER Software für Computational Clearing. Das Mikroskop ist programmierbar für komplexe Aufgaben, wie beispielsweise das Abscannen von Multiwellplatten. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Lebendzellmikroskopie, Weitfeldmikroskopie, Phasenkontrastmikroskopie, DIC, Scannen von Multiwellplatten

Foto: UHH/Feuerböther
Leica DM6 B aufrechtes Weitfeldmikroskop #1
Beschreibung: Dieses aufrechte Weitfeldmikroskop von Leica ist ausgestattet mit einer Fluoreszenzanregungslichtquelle. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Weitfeldmikroskopie, Phasenkontrastmikroskopie, DIC, Histologie

Foto: UHH/Feuerböther
Leica DM6 B aufrechtes Weitfeldmikroskop #2
Beschreibung: Dieses aufrechte Weitfeldmikroskop von Leica ist ausgestattet mit einer Fluoreszenzanregungslichtquelle und ist voll motorisiert. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Weitfeldmikroskopie, Phasenkontrastmikroskopie, DIC, Histologie

Foto: UHH/Feuerböther
Leica DM6 B aufrechtes Weitfeldmikroskop #3
Beschreibung: Dieses aufrechte Weitfeldmikroskop von Leica ist ausgestattet mit einer Fluoreszenzanregungslichtquelle. Das Mikroskop im BSL2 Labor von Tim Gilberger am CSSB Hamburg untergebracht.
Hauptanwendungen: Weitfeldmikroskopie, Phasenkontrastmikroskopie, DIC, Histologie

Foto: UHH/Feuerböther
Leica SR GSD 3D Super-Resolution-Mikroskop
Beschreibung: Dieses inverse Mikroskop von Leica ist ausgestattet für Single Molecule Localization Microscopy (SMLM) Super-Resolution Mikroskopie und einer speziellen Stage, die direkt an das Objektiv gekoppelt ist. Die Fluoreszenzanregung kann im Weitfeldmodus und im Total Internal Reflection Fluorescence (TIRF)-Modus erfolgen. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Super-Resolution Mikroskopie, TIRF, STORM, DNA-PAINT

Foto: UHH/Feuerböther
Spinning-Disc- und SIM-Mikroskop mit FRAP Einheit
Beschreibung: Dieses inverse Mikroskop von Nikon hat mehrere Modi in denen es betrieben werden kann. Es ist eine Yokogawa CSU W1 Spinning Disc Einheit mit einer Andor iXon EMCCD Kamera vorhanden und eine Nikon E-SIM Einheit für Structured Illumination Microscopy (SIM). Darüber hinaus ist das Mikroskop mit einer Einheit für Fluorescence Recovery after Photobleaching (FRAP) von Rapp Optoelectronics ausgestattet. Es ist eine Inkubationseinheit für Lebendzellmessungen vorhanden. Das Mikroskop ist voll programmierbar über das JOBS-Modul in der Nikon NIS Elements Software. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Spinning-Disc-Konfokalmikroskopie, SIM, FRAP, Photoaktivierung, Lebendzellmikroskopie, Scannen von Multiwellplatten

Foto: UHH/Feuerböther
LSM880 Airyscan Konfokalmikroskop
Beschreibung: Dieses inverse Point-Scanning-Konfokalmikroskop von Zeiss hat einen Airyscandetektor, welcher genutzt werden kann, um eine ungefähr 1,8-fache Erhöhung der Auflösung gegenüber normalen Konfokalmikroskopen zu erreichen, und einen Spectral Imaging Detektor. Es ist mit einer Inkubationskammer für Lebendzellmessungen ausgestattet. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Konfokalmikroskopie, Lebendzellmikroskopie, Airyscan-Mikroskopie

Foto: UHH/Feuerböther
Elyra PS.1 Super-Resolution-Mikroskop
Beschreibung: Dieses inverse Super-Resolution-Mikroskop von Zeiss kann für Single Molecule Localization Microscopy (SMLM) Mikroskopie und für Structured Illumination Microscopy (SIM) verwendet werden. Die Fluoreszenzanregung kann im Weitfeldmodus oder im Total Internal Reflection Fluorescence (TIRF)-Modus erfolgen. Es ist mit einer Inkubationskammer für Lebendzellmessungen ausgestattet. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Super-Resolution Mikroskopie, TIRF, STORM, PALM, DNA-PAINT, SIM, Weitfeldmikroskopie

Foto: UHH/Feuerböther
Lattice-Light-Sheet-Mikroskop
Beschreibung: Das Lattice-Light-Sheet-Mikroskop von Zeiss ist schnelle volumetrische Aufnahmen von lebenden Zellen mit besonders geringer Phototoxizität und gleichzeitig hoher örtlicher Auflösung geeignet. Es verfügt über zwei Hamamatsu sCMOS Kameras. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Lattice-Light-Sheet Mikroskopie, Lebendzellmikroskopie

Foto: UHH/Feuerböther
BSL2 Zellkulturwerkbank und Inkubatoren
Beschreibung: Die Zellkulturwerkbank und zwei Inkubatoren sind voll ausgestattet damit Nutzende der ALFM-Facility ihre Lebendzellproben für die Mikroskopie vorbereiten und vorinkubieren können. Die Zellkulturwerkbank und die Inkubatoren sind in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Vorbereitung von Lebendzellproben für die Mikroskopie

Foto: UHH/Feuerböther
Leica M125C Stereomikroskop
Beschreibung: Das M125C ist ein kleines Stereomikroskop von Leica mit LED-Weißlichtquelle. Das Mikroskop ist in einem Labor der biologischen Sicherheitsstufe 2 (BSL2) untergebracht.
Hauptanwendungen: Inspektion und Präparation von Mikroskopieproben.

Foto: UHH/Feuerböther
Analysecomputer (Platzhalter-Bild)
Beschreibung: Der Analysecomputer verfügt über 768 GB Arbeitsspeicher und zwei NVIDIA RTX A6000 Graphikkarten für die Bearbeitung und Analyse großer Bilddatenmengen. Auf dem Computer sind FIJI, Imaris, Arivis und Matlab als Bildanalysesoftware vorhanden.
Hauptanwendungen: Bildbearbeitung und quantitative Bildanalyse von Mikroskopiedaten
