8. August 2023
Antibiotika-Ribosom-Verbindungen entschlüsseltBekämpfung multiresistenter Bakterien durch hochauflösende Strukturdarstellungen
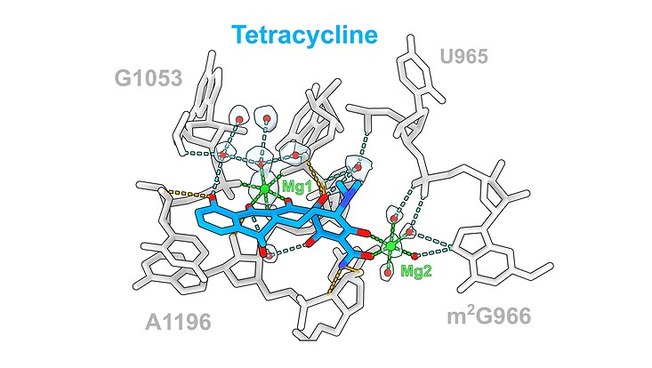
Foto: UHH/Paternoga
Ribosomen sind große Molekülkomplexe, die in den Zellen von Pflanzen, Tieren, Menschen sowie Bakterien vorkommen und Proteine herstellen. Deshalb sind sie wichtige Ziele für Antibiotika, die bei Bakterien an eine Untereinheit der Ribosomen ankoppeln und Ablesefehler oder gar Ablesestopps verursachen. Dadurch werden fehlerhafte Proteine gebildet, die ihre biologische Funktion verlieren, und das Bakterium stirbt. Allerdings können mehr und mehr multiresistente Bakterien die Wirkung von Antibiotika verhindern.
Um diese Resistenzen zu umgehen, ist das genaue Verständnis der Antibiotika unerlässlich, insbesondere der Antibiotika-Ribosom-Strukturen. In den vergangenen zwei Jahrzehnten wurden Antibiotika-Ribosom-Strukturen für jede wichtige Klasse Ribosom-gerichteter Antibiotika veröffentlicht, die Einblicke in ihre Bindungsstellen und Wirkmechanismen geben.
Das Team um Prof. Dr. Daniel N. Wilson vom Fachbereich Chemie der Universität Hamburg hat diese Strukturen nun mithilfe von Kryo-Elektronenmikroskopie mit einer Auflösung von 1,6 bis 2,2 Ångström (Å) dargestellt, wobei ein Å dem zehnmillionsten Teil eines Millimeters entspricht. „In der Vergangenheit wurden hauptsächlich Antibiotika-Ribosom-Strukturen mit Auflösungen von 2,5 bis 3,5 Å präsentiert“, sagt Prof. Dr. Daniel N. Wilson. „Die verbesserte Auflösung ermöglicht uns nun, sogar Wassermoleküle zu beobachten, welche die Wechselwirkungen zwischen den Medikamenten unterstützen.“ Die Forschenden fanden heraus, dass im Allgemeinen zehn bis 20 Wasserstoffbindungen mit dem Ribosom interagierten, wobei der Beitrag der Wassermoleküle für die verschiedenen Antibiotikaklassen unterschiedlich ist.
Die untersuchten Antibiotika umfassen die sechs klinisch relevanten Antibiotikafamilien, darunter die Tetracycline, die bei Atemwegsinfektionen zum Einsatz kommen, Aminoglycoside mit einem breiten Wirkungsspektrum, unter anderem bei Tuberkulose oder Meningitis, sowie die Pleuromutiline gegen Haut- und Weichgewebsinfektionen.
Für die Studie setzte das Forschungsteam diese Antibiotika auf sogenannte 70S-Ribosomen des Bakteriums „Escherichia coli“ an – ein Kolibakterium, das sich auch im menschlichen Darm befindet. „Wir gehen davon aus, dass diese Informationen in Zukunft für das strukturbasierte Design neuer Antibiotikaderivate genutzt werden können. Indem wir Regionen identifizieren, die verändert werden können, kann das gebundene Wasser gezielt verdrängt werden und so eine Wechselwirkung mit dem Ziel, also dem Bakterium, übernehmen“, sagt Wilson.
Die hochaufgelöste Darstellung ist Wilson zufolge ein großer Schritt: „Die alten Antibiotika-Ribosom-Strukturen mit weniger hoher Auflösung zeigen, dass die Bindungsstellen für jede Antibiotika-Klasse ähnlich sind, allerdings gibt es in vielen Fällen tiefgreifende Unterschiede in Bezug auf die genaue Position der Wirkstoffe sowie der Wirkstoffbindungsstelle“. Daher seien die hochauflösenden experimentellen Daten benötigt worden, um eine genauere Beschreibung der Interaktionen von Antibiotika mit dem Ribosom zu erhalten.
An der Studie waren neben der Universität Hamburg auch Forschende des Max-Planck-Instituts für Multidisziplinäre Naturwissenschaften in Göttingen, des Dubochet Center for Imaging at EPFL-UNIL in der Schweiz und des Central European Institute of Technology (CEITEC) in der Tschechischen Republik beteiligt.
Originalpublikation:
Paternoga, Crowe-McAuliffe, Bock, Koller, Morici, Beckert, Myasnikov, Grubmüller, Nováček, Wilson (2023) Structural conservation of antibiotic interaction with ribosomes, Nature Structural & Molecular Biology.
https://doi.org/10.1038/s41594-023-01047-y
