Mit Computersystemen auf der Suche nach Wirkstoffen„Wir beschreiben Moleküle wie einen Legobausatz“
18. Mai 2020, von Maria Latos
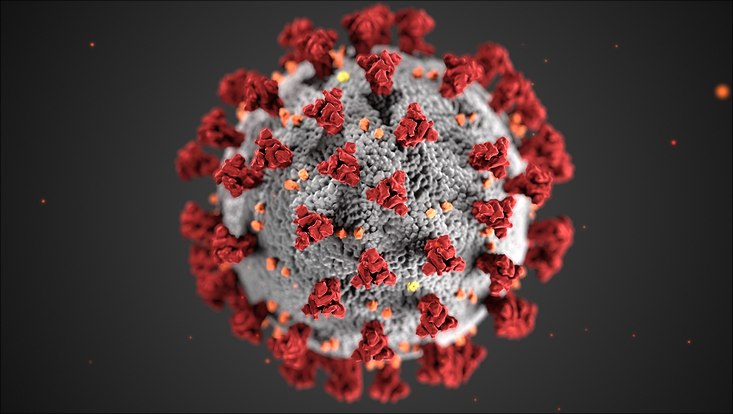
Foto: CDC/ Alissa Eckert, MS; Dan Higgins, MAM
Derzeit wird unter Hochdruck nach einem Wirkstoff gegen das Coronavirus gesucht. Professor Dr. Matthias Rarey, Leiter der Forschungsgruppe für Algorithmisches Molekulares Design am Zentrum für Bioinformatik (ZBH) der Universität Hamburg, und seine wissenschaftliche Mitarbeiterin, Dr. Christiane Ehrt, erklären, wie die Bioinformatik bei der Wirkstoffsuche helfen kann.
Professor Rarey, Sie sind mit Ihrer Forschungsgruppe auch in der Wirkstoffforschung tätig. Was untersuchen Sie?
Rarey: Die Entwicklung eines Medikaments ist ein sehr langwieriger Prozess. Typischerweise dauert es zehn Jahre, bis ein neues Mittel auf den Markt kommt. Bei einem Wirkstoff gegen ein Virus steht am Anfang die sogenannte Target-Identifizierung: Es wird untersucht, an welchem Zielprotein des Virus eine Behandlung angesetzt werden könnte. In der zweiten Phase der Wirkstoffentwicklung will man herausfinden, welche Moleküle für diese Zielproteine als potenzieller Wirkstoff in Frage kommen. Wir werden in dieser zweiten Phase aktiv.

Wie erforschen Sie das?
Rarey: Es gibt sehr viele organische Verbindungen und damit viele mögliche Wirkstoffe. Wir suchen also die berühmte Nadel im Heuhaufen. Wir entwickeln und nutzen Computersysteme, die uns bei der Suche und bei der Vorhersage, wie die Moleküle an die Zielproteine binden, helfen.
Ehrt: Der Großteil der Arbeitsgruppe widmet sich der Entwicklung der Softwaresysteme, die international eingesetzt werden. Mit einigen Kollegen wende ich die entwickelten Methoden aber auch an, um aktuelle Fragestellungen zu bearbeiten.
Was sind das für Softwaresysteme?
Rarey: Wir entwickeln Modelling-Verfahren auf sogenannten „chemischen Fragmenträumen“. Dabei beschreiben wir die Moleküle wie einen Legobausatz. Das heißt, wir haben Komponenten, die wie einzelne Legosteine sind und wir können mit unseren Computerverfahren präzise beschreiben, welche wie miteinander kombiniert werden können. Mit unseren Softwaresystemen konstruieren wir die Moleküle also maßgeschneidert für ausgewählte Zielproteine. So können wir Moleküle entwerfen, die potenziell an die Proteine binden und die dann auch synthetisch im Labor herstellbar sind.
Und diese Methodik setzen Sie gerade für die Wirkstoffsuche gegen das Coronavirus ein?
Rarey: Die Strategie ist in dem Fall etwas anders. Es nützt heute wenig, wenn wir einen organische Verbindung vorschlagen, die vielleicht in zehn Jahren ein Wirkstoff wird. Wir suchen deshalb zielgerichtet in Bibliotheken von synthetischen Molekülen, die bereits für den Einsatz im Menschen optimiert und teilweise am Menschen getestet sind. Es geht bei uns um das „drug repurposing“, also die Verwendung eines existierenden Wirkstoffs für einen neuen Anwendungszweck.
Ehrt: Es geht hauptsächlich um bekannte Zielproteine von SARS-CoV-2. Da haben wir schon einige Kenntnisse, zum Beispiel was deren atomare Struktur angeht. Für diese Targets können wir mit verschiedenen Methoden der Arbeitsgruppe in Moleküldatenbanken suchen und schauen, ob es möglich ist, dass bereits bekannte Wirkstoffe an das Zielprotein binden können.

Und wenn sie das können?
Ehrt: Dann sind diese Moleküle hochinteressante Ausgangspunkte für experimentelle Studien, die zu Therapien zur Behandlung von COVID-19 führen könnten.
Haben Sie denn schon Wirkstoffe in den Moleküldatenbanken gefunden?
Ehrt: Wir sind derzeit noch in einer Validierungsphase. Das ist übrigens eine parallele Suche. Wir versuchen mit computerbasierten Ansätzen herauszufinden, welcher Wirkstoffkandidat in einer Datenbank interessant wäre. Unsere Kolleginnen und Kollegen am DESY untersuchen gleichzeitig mithilfe experimenteller Ansätze die gleiche Molekülbibliothek und versuchen auf diese Weise mögliche Kandidaten zu finden.
Rarey: Es ist ein Wechselspiel: Wir bekommen von den Arbeitsgruppen des DESY die Strukturinformationen von Molekülen und liefern auf der anderen Seite wieder Vorschläge für Wirkmechanismen. Und mittelfristig wollen wir natürlich auch Vorschläge für neue Moleküle entwickeln, die man testen könnte.
Braucht es also neben den computergestützten Methoden und Ansätzen auch immer die experimentellen Methoden?
Rarey: Man kann es mit einer Wettervorhersage vergleichen: Wir können eine Vorhersage machen, die natürlich nur eine gewisse Genauigkeit hat. Am Ende sind es die experimentellen Methoden, welche die finalen Ergebnisse liefern. Aber bei den verschiedenen Schritten hilft die Bioinformatik mit ihren Spezialisierungen. Denn auch im Bereich der Sequenzierung des Virus-Erbmaterials sowie bei der Erstellung von Strukturmodellen der Moleküle ist die Fachrichtung gefragt.
Wie zum Beispiel das Bild des Coronavirus, das man überall in den Medien sieht?
Rarey: Genau, hier setzt die strukturelle Bioinformatik an. Dazu gehört sowohl das Bauen dieser Modelle als auch das Arbeiten damit. In dieses Modell kann man bis auf die atomare Ebene heranzoomen, bis zu jedem einzelnen Atom des Spike-Proteins - das sind die Fortsätze, die man außen sieht. Und das sind genau die Modelle, die wir in der Wirkstoffforschung brauchen, weil wir auf atomarer Ebene arbeiten und untersuchen, wie die Moleküle miteinander interagieren. Es geht dann weiter in die Dynamik, also die Frage, wie sich die Moleküle bewegen. Die Bilder in den Medien suggerieren ja, dass das Virus eine starre Kugel wäre. Aber das Virus hat natürlich eine Dynamik, es bewegt sich. Diese Dynamik der Proteine ist häufig entscheidend für ihre Funktionen und man bildet die Bewegung der Atome in diesen Strukturen nach, um ein Verständnis für die funktionellen Abläufe zu bekommen.
Weitere Wirkstoffforschung
Wie am Exzellenzclusters CUI nach neuen Wirkstoffen gesucht wird, ist in dieser Pressemitteilung sowie im dazugehörigen Interview zu lesen.
Masterstudium Bioinformatik
Der Fachbereich Informatik bietet den Masterstudiengang Bioinformatik an. Hier werden in dem zweijährigen, forschungsorientierten Studiengang die Wissensgebiete Lebenswissenschaften und Informatik kombiniert. Im Rahmen des Studiums besteht die Möglichkeit der Spezialisierung in Genominformatik, Strukturelle Bioinformatik und Chemieinformatik/Wirkstoffentwurf.


